- Mikaël Salson
- Professeur en informatique
- mikael.salson@univ-lille.fr
- +33 3 28 77 84 65
- Prendre rendez-vous : En personne / À distance
Parcours
- Je fais partie depuis septembre 2010 de l'équipe de recherche en bioinformatique Bonsai (CRIStAL – Université de Lille et CNRS). Vous pourriez me trouver dans les environs du bureau S3.57 dans le bâtiment ESPRIT ou de celui du bureau 216 de l'extension du bâtiment M3, sur le chemin entre les deux ou à tout autre endroit.
- J'ai effectué ma thèse à l'université de Rouen au sein de l'équipe TIBS du laboratoire LITIS.
Enseignement
- Démarche scientifique et esprit critique (responsable)
- Licence 3è année – Informatique 2014 →
- Journalisme - Sciences Po Lille 2022 →
- Master 2è année – Journaliste et scientifique 2013–2022
- Algorithmes et structures de données (responsable)
- Licence 2è année – Informatique 2020 →
- Compression de l'information détection et correction d'erreurs (responsable)
- Licence 2è année – Informatique 2020 →
- Initiation à la programmation
- Licence 3è année – Biologie des organismes et des populations (BOP) 2021 →
- Bioinformatique
- Licence 3è année – Informatique 2020 →
- Méthodes d'analyses bioinformatiques des séquences
- Master 1è année – Bioinformatique (parcours MISO) 2020 →
- Bases de données
- Master 1è année – Bioinformatique (parcours MISO) 2022 →
Recherche
Je suis responsable de l'équipe de recherche Bonsai.
Domaines de prédilection
Indexation de séquences
Le volume de données de séquençage disponible rend nécessaire l'indexation des données, que ce soit au niveau d'un génome, d'un jeu de séquençage ou de collections de jeux de séqeuençage. Ces différentes facettes de l'indexation visent un même but : accéder rapidement aux données pour en tirer parti au mieux. Plusieurs travaux de thèses ou post-doctorats que j'ai co-encadrés ont abordé ces aspects :/
-
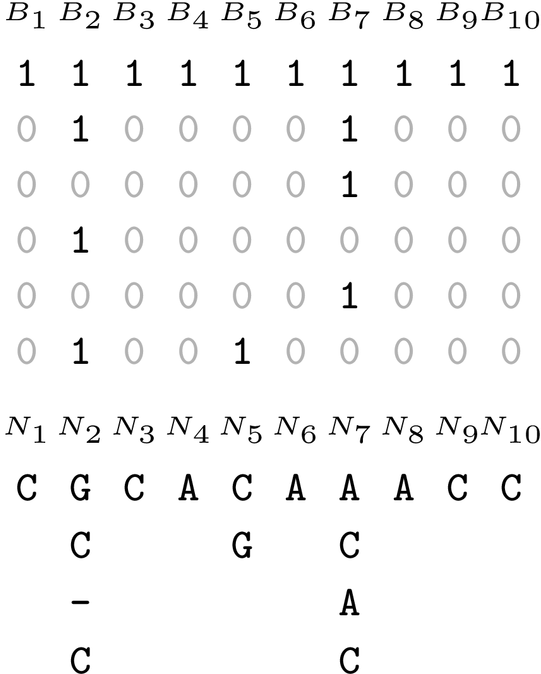
Indexation d'alignement multiple de séquences afin de calculer efficacement des statistiques de covariation sur des colonnes de l'alignement multiple.
-
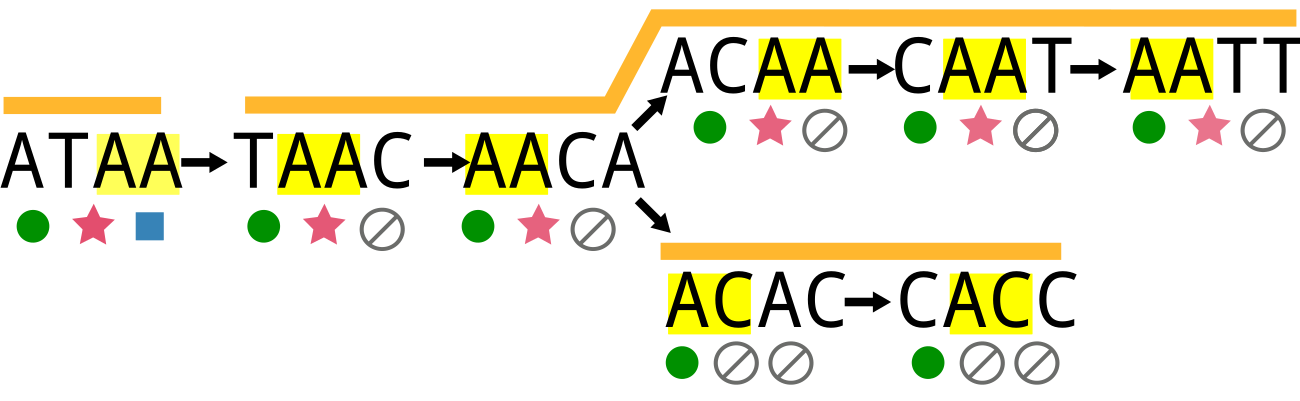
Indexation de jeux de données de séquençage (post-docs de C. Marchet et F. Ingels)
-
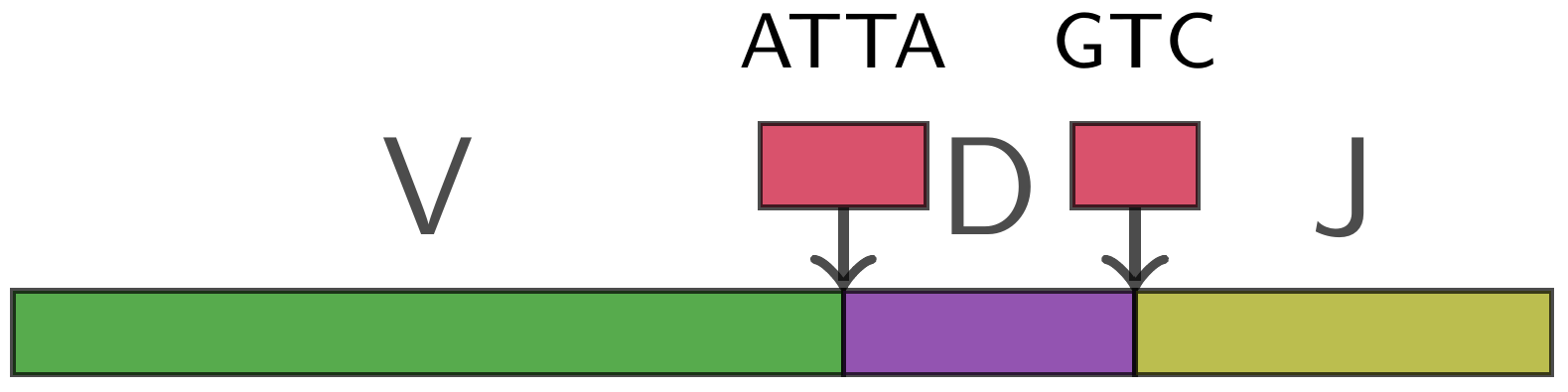
Indexation de recombinaisons V(D)J (thèse de T. Rocher)
-
Indexation de génomes pour la recherche de courtes séquences avec de fort taux d'erreurs (appliquée aux cibles de micro-ARN) (thèse de C. Vroland)
Comparaison sans alignement
Les comparaisons sans alignement sont des approches qui comparent de données sans procéder à de l'alignement par programmation dynamique. Ces approches ont le mérite d'être beaucoup plus frugales sans nécessairement perdre en qualité (contrairement à ce qui pourrait être craint).
-
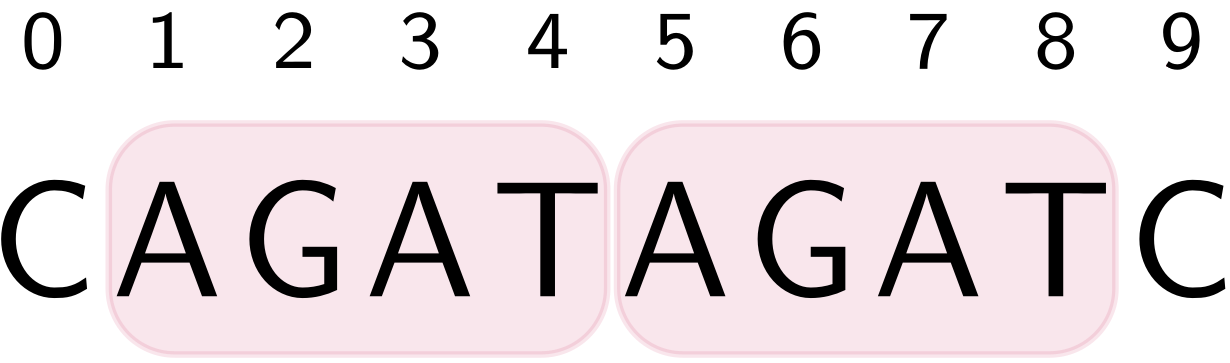
Identification de duplications en tandem dans des gènes connus (application à la recherche de FLT3-ITD dans les leucémies aiguës myéloïdes)
-
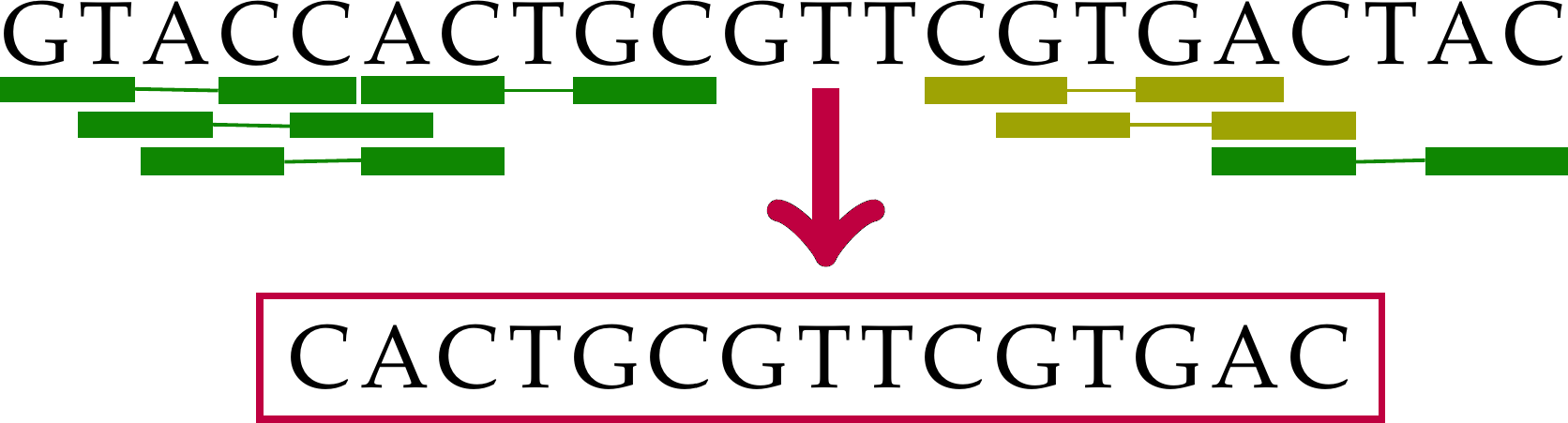
Caractérisation des populations de globules blancs par leurs recombinaisons V(D)J (application à l'identification de marqueurs dans les leucémies)
-
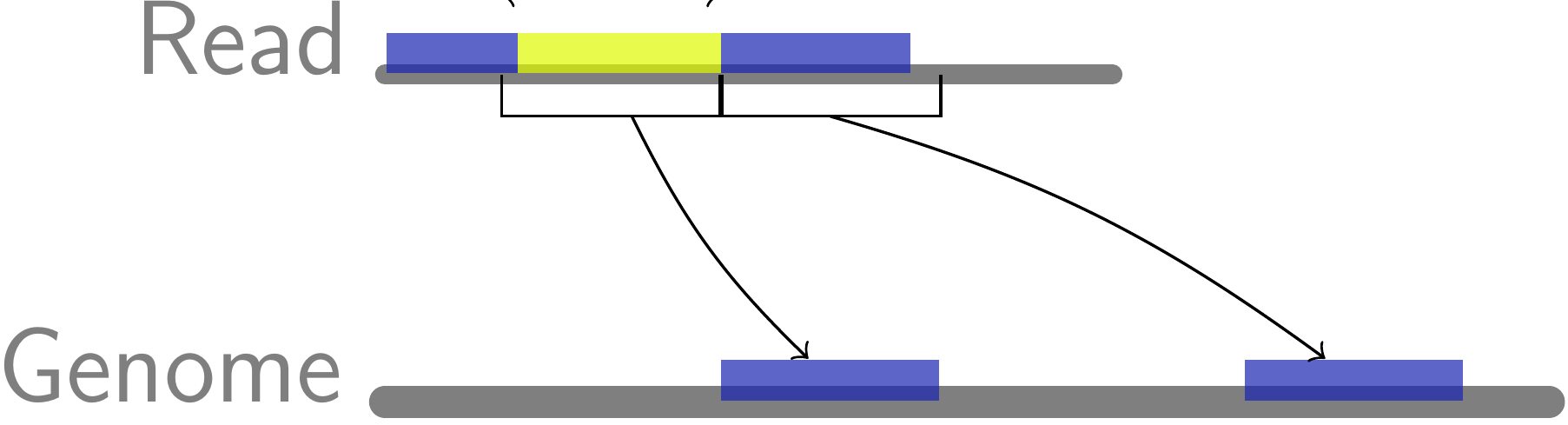
Analyse de données de RNA-seq (détection d'épissages, fusions, etc.)
Par ailleurs je co-encadre la thèse de T. Baudeau sur l'analyse de séquençage Nanopore de virus.
Logiciels
- CREMSA
- Logiciel pour l'indexation de très grands alignements multiples de séquences.
- FiLT3r
- Logiciel pour l'analyse de duplications en tandem dans un ou plusieurs gènes d'intérêt à partir de données de séquençage à haut débit. Le logiciel a été initialement développé pour la recherche des FLT3-ITD mais peut être utilisé plus généralement.
- Vidjil
- Une plateforme logicielle pour l'analyse de populations de lymphocytes, à partir de leurs recombinaisons V(D)J, dans des données de séquençage à haut-débit. Cette plateforme est notamment utilisée pour le suivi de la leucémie par plusieurs hôpitaux dans le monde. Le logiciel est désormais transféré au sein du consortium sans but lucratif VidjilNet
- CRAC
- Un outil d'analyse de données RNA-Seq : détection de jonctions d'épissage (alternatif ou non), de gènes ou transcrits de fusion, de mutations ponctuelles (substitutions, insertions, délétions).
- GkArrays
- Bibliothèque C++ pour l'indexation des lectures issues des séquenceurs haut-débit.
- DFMI
- Implantation d'une structure d'indexation dynamique et compressée.
Publications
Présentations données
Conflits d'intérêt
Je possède le nombre minimal de parts dans ma banque coopérative. Mes recherches sont conduites à l'aide de fonds publics exclusivement. Dans le cadre du projet Vidjil, j'ai travaillé en 2015–2016 avec un ingénieur financé par la fondation EDF. J'ai signé une convention d'orateur non rémunéré dans le cadre de deux journées organisées par Novartis à Dieppe en 2016 sur l'onco-hématologie avec des médecins de différents CHU du nord de la France. La nuit d'hôtel et trois repas ont été pris en charge par Novartis. En janvier 2020 j'ai été invité à Montpellier par l'entreprise SeqOne qui a pris en charge le trajet et quelques repas. Au printemps 2020 j'ai effectué une mission d'expertise pour cette même entreprise. Les fonds provenant de cette mission d'expertise ont été attribués à mon laboratoire, qui ont été utilisés pour financer un stagiaire, une publication et un repas d'équipe.
Contact
Université de Lille – CRIStAL
Bâtiment M3 – Cité scientifique
59655 Villeneuve d'Ascq Cedex - France
+33 3 28 77 84 65

Crédits : Image de l'indexation de jeux de données de séquençage par Camille Marchet, image de la tige-boucle CC BY SA par Sakurambo.
